SPLICING DE INTRONES
INTRODUCCIÓN:
En los procariontes, los
ribosomas se unen a una molécula de ARNm en crecimiento y su traducción a una
proteína comienza aun antes de que se haya completado la transcrpción. A diferencia
de los anteriores la transcripción y la traducción de los eucariontes se
encuentran separadas en el tiempo y en el espacio. En la transcripción de los
eucariotas están implicadas tres ARN polimerasas diferentes, cada una
especializada en transcribir distintos tipos de genes. Además, se requieren
factores generales de transcripción, que permiten la unión de las ARN
polimerasas al promotor, así como una multiplicidad de proteínas regulatorias.
Además, en los eucariotas los genes estructurales no están agrupados en
operones como lo están frecuentemente en los procariotas; la transcripción de
cada gen se regula por separado y cada gen produce un transcripto de ARN que
contiene la información codificada de un solo producto. Una vez que el núcleo
se ha completado la transcripción por medio de la ARN polimerasa II, los
transcriptos de ARNm (ARNm inmaduro o primario) se terminan de procesar
modificando los extremos logrando las moléculas maduras antes de ser
transportados al citoplasma celular a través de los poros nucleares. Este
procesamiento incluye la adición
DESARROLLO;
El splicing de ARN o empalme
de ARN es un proceso post-transcripcional de maduración del ARN del cual
eliminan ciertos fragmentos secuenciales. Este proceso es muy común en
eucariotas, pudiéndose dar en cualquier tipo de ARN aunque es más común en el
ARNm. También se ha descrito en el ARNr y ARNt de procariotas y bacteriófagos.
Normalmente consiste en eliminar los intrones del transcrito primario y
posteriormente unir los exones; aunque existen otros tipos de ajuste donde se
eliminan exones y/o retienen intrones.
RUTAS DE SPLICING
En la naturaleza existen
diversos métodos de splicing del ARN. El mecanismo de splicing depende de la estructura
del fragmento de ARN que pasará por este proceso.
SPLICEOSOMA:
El Spliceosoma es un
complejo formado por cinco ribonucleoproteínas nucleares pequeñas o snRNP
(complejo formado por unas diez proteínas más una pequeña molécula de ARN). El
ARN de los snRNP es el encargado de reconocer el intrón. Se han identificado
dos tipos de spliceosomas, el mayor y el menor[, cada uno de los cuales
contiene diferentes tipos de snRNP.
SPLICEOSOMA MAYOR
Está formado por los snRNP
U1, U2, U4, U5 y U6. Reconoce la secuencia consenso GU (Guanina-Uracilo) del
extremo 5’ del intrón así como la secuencia consenso AG del extremo 3’. El 99%
de los intrones lo hacen a través de este mecanismo.
*Complejo E: U1 se une a la
secuencia consenso GU del extremo 5’ del sitio de corte del intrón, junto con
las proteínas accesorias ASF/SF2, U2AF, SF1/BBP.
*Complejo A: U2 se une al
sitio de ramificación e hidroliza ATP. El sitio de ramificación se sitúa a una
distancia de 20-40 nucleótidos del extremo 3’ del intrón y en él se localiza la
secuencia consenso CURAY.
*Complejo B1: U5, U4 y U6
trimerizan, y U5 se une al exón 5’ y U6 a U2.
*Complejo B2 – U1 es
liberado, U5 pasa del exón al intrón y U6 se une al extremo 5’ del sitio de
corte.
*Complejo C1: U4 es
liberado, U5 se une al sito de empalme del extremo 3’ del exón, U6 y U2
catalizan la reacción de transesterificación y el extremo 5’ del intrón es
cortado; como resultado se forma una estructura en lazo característica denominada
lariat.
*Complejo C2: el extremo 3’
del intrón es cortado lo que provoca la liberación del lazo de ARN. A
continuación los exones son ligados, lo que conlleva gasto de ATP. Por último,
el complejo se disocia.
SPLICEOSOMA MENOR
Es similar al Spliceosoma
mayor aunque los intrones eliminados mediante este mecanismo son escasos, y
además presentan diferencias en los sitios de corte y empalme. También se
diferencian en las secuencias consenso reconocidas, que en este caso son AU y
AC para los extremos 3’ y 5’, respectivamente. Además, salvo la partícula snRNP
U5, el resto son análogos funcionales denominadas U11 (análogo funcional de la
U1), U12 (U2), U4atac (U4) y U6atac(U6).
En la mayoría de los casos,
el ARN mensajero sufre la eliminación de secuencias internas, no codificantes,
llamadas intrones. Esto no ocurre en células procariontes, ya que estas no
poseen intrones en su ADN. El proceso de retirada de los intrones y conexión o
empalme de los exones se llama ayuste, o corte y empalme (en inglés, splicing).
A veces un mismo transcrito primario o pre-ARNm se puede ayustar de diversas
maneras, permitiendo que con un solo gen se obtengan varias proteínas
diferentes; a este fenómeno se le llama ayuste alternativo. Ciertas enzimas
parecen estar involucrados en editar el RNA antes de su exportación fuera del
núcleo, intercambiando o eliminando nucleótidos erróneos. Por esto, es posible
decir, que el plegamiento que sufre el ARNm momentos antes de la eliminación de
los intrones, le confiere una estructura secundaria que a su vez, perderá en el
momento en el que esos intrones, sean eliminados.
PROCESO:
Se trata de un mecanismo muy
exacto, pues de no serlo produciría un corrimiento del marco de lectura en el
mensaje transcripto. Los intrones son cortados del ARNm inmaduro por un sistema
específico que reconocen secuencias cortas dentro de él y que se encuentra
cerca de los límites con exones. Estas secuencias son llamadas "sitio
dador" (común en casi en todos los intrones), en el extremo 5’y
"sitio aceptor", en el extremo
El splicing es el proceso de eliminación de intrones y unión de exones durante la maduración de los pre-RNAs.
El trabajo del corte y
empalme esta catalizado por una estructura pequeña, compuesta por
ribonucleoproteinas nucleares llamadas snRNPs, constituidas por pequeños ARN
nucleares (snARNs) asociado a proteínas. Su nombre es spliceosoma. Esta estructura
tiene a su cargo el reconocimiento de las secuencias mencionadas anteriormente
en los intrones y su posterior fijación. Luego se desarrollan una secuencia de
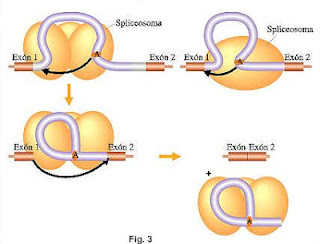
pasos que determinan el clivaje y ligado de los intrones y exones (Fig. 3):
1 el extremo 5’del intrón es
clivado y unido a otros sitio interno del intrón, cercano a su extremo 3’
llamado "sitio de ramificación" .
2 Se produce el corte en el
extremo 3’ del intrón y son empalmados los dos exones de cada lado, liberándose
el ARNm maduro del spliceosoma.
3 El intrón eliminado queda
formando una estructura con forma de lazo, llamada"lariat", que
posteriormente es degradado en el núcleo.
Se ha observado que ARNm
inmaduros idénticos del mismo gen se procesan en más de una forma. Esto
significa que existen diversos empalmes alternativos, los cuales desarrollaran
diversos ARNm maduros y por lo tanto distintos polipéctidos funcionales.
VÍAS DE EMPALME O SPLICING
Varios métodos de empalme de
ARN se producen en la naturaleza, el tipo de empalme depende de la estructura
de la empalmados intrón y los catalizadores necesarios para el empalme que se
produzca.
SPLICEOSOMAL INTRONES
Intrones spliceosomal menudo
residen dentro de la secuencia de eucariotas codificación de proteína de los
genes. Dentro del intrón, sitio de empalme, de 5' un sitio de empalme 3, y el
sitio de la rama se requieren para el empalme. El 5 'del sitio de empalme o en
el sitio donador de empalme incluye una secuencia casi invariable GU en el
extremo 5' del intrón, dentro de una más grande, región consenso menos
altamente conservada. El sitio de la 3 'de empalme o en el sitio aceptor de
empalme del intrón termina con una secuencia de AG casi todos los idiomas.
Aguas arriba (5'-Ward) de la AG hay una región de alta en pirimidinas (C y T),
o tracto polipirimidina .
Aguas arriba de la zona polypyrimidine es el punto de
ramificación, que incluye una adenina nucleótidos.Las mutaciones puntuales en
el ADN subyacente o errores durante la transcripción se puede activar un
"sitio de empalme críptico" en la parte de la transcripción que
normalmente no se empalma. Esto da como resultado un mensajero maduro ARN con
una sección que falta de un exón. De esta forma un punto de mutación , que
generalmente sólo afecta a un solo aminoácido, puede manifestarse como una
dilección en la proteína final.
EMPALMOSOMA LA FORMACIÓN Y LA
ACTIVIDAD
Empalme es catalizada por la
spliceosome que es un gran ARN-proteína compuesta por cinco ribonucleoproteínas
pequeñas nucleares ( snRNPs , que se pronuncia 'snurps'). Los componentes del
RNA de snRNPs interactuar con el intrón y puede estar implicada en la
catálisis. Hay dos tipos de spliceosomas se han identificado (el mayor y
menor), que contienen diferentes snRNPs .
·
Mayor
Los intrones que contienen
los principales empalmes spliceosome GU en el 'sitio de empalme y AG en el
extremo 3' del sitio de empalme 5. Se compone de la U1 , U2 , U4 , U5 , y U6
snRNPs y está activa en el núcleo. Además, un número de proteínas que incluyen
U2AF y SF1 se requieren para el montaje de la spliceosome.
§ E-U1 complejo se une a la secuencia GU en el
sitio de empalme 5 ', junto con proteínas accesorias y enzimas ASF/SF2, U2AF
(se une en el sitio de Py-AG), SF1/BBP (BBP = Proteína Poder atar);
§ Un complejo-U2 se une a la sucursal y el ATP
se hidroliza;
§ B1 Complex-U5/U4/U6 trímero se une, y la U5
se une los exones en el sitio 5 ', con U6 vinculante a U2;
§ Complejo B2-U1 es liberado, U5 cambios desde
el exón a intrón y el U6 se une al sitio de empalme 5 ';
§ C1 Complejo-U4 es liberado, U6/U2 cataliza la
transesterificación, que hacen extremo 5 'de los intrones ligate a la A en el
intrón y formar un lazo, U5 se une a exón 3' del sitio de empalme, y el 5 'del
sitio escindido se, lo que resulta en la formación del lazo;
§ C2 Complex-U2/U5/U6, permanece vinculado a la
reata, y el sitio 3 'se escinde y los exones se ligan mediante la hidrólisis de
ATP. El empalmados ARN se libera y los debranches lariat.
Este tipo de empalme que se
denomina empalme canónico o llama la vía de lazo, que representa más del 99% de
empalme. Por el contrario, cuando las secuencias intrónicas flanqueantes no
siguen la regla GU-AG, el empalme no canónico se dice que ocurre.
Menor de edad
El spliceosome menor es muy
similar a la spliceosome importante, sin embargo se empalma a cabo intrones
raras con diferentes secuencias de sitio de empalme. Mientras que los
spliceosomas menores y mayores contienen el mismo U5 snRNP , el spliceosome
menor tiene diferente, pero funcionalmente análogos para snRNPs U1, U2, U4 y
U6, que se llama, respectivamente,U11 , U12 , U4atac y U6atac. Al igual que el spliceosome importante, sólo
se encuentra en el núcleo.
· Trans-empalme
Trans-empalme es una forma
de empalme que une dos exones que no están dentro del mismo transcrito de ARN.
AUTO-EMPALME
Auto-empalme de intrones
ocurre raras que forman una ribozima , la realización de las funciones de la
spliceosome de ARN solo. Hay tres clases de intrones de empalme de sí mismo, el
Grupo I , Grupo II y Grupo III . Grupo I y II intrones realizar el empalme
similar a la spliceosome sin requerir ninguna proteína. Esta similitud sugiere
que intrones del grupo I y II pueden ser evolutivamente relacionados con el
spliceosome. Auto-empalme también puede ser muy antiguo, y puede haber existido
en un mundo de ARN presentes antes de la proteína. Aunque los dos mecanismos de
empalme se describe a continuación no requieren ninguna proteína que se
produzca, 5 adicionales moléculas de ARN y más de 50 proteínas y moléculas se
utilizan muchos hidroliza ATP. Los mecanismos de empalme utiliza ATP con el fin
de empalmar con precisión de ARNm. Si la celda eran de no usar ninguna de la
ATP, el proceso sería muy inexacto y muchos errores se produciría.
Dos transesterificaciones
caracterizar el mecanismo en el que intrones del grupo I se empalman:
1. 3'OH de un libre de nucleósido de guanina
(o uno ubicado en el intrón) o un cofactor de nucleótidos (GMP, GDP, GTP) ataca
el fosfato en el sitio de empalme de los 5’.
2. 3'OH del 5'exon se convierte en un
nucleófilo y los resultados de transesterificación segundo en la unión de los
dos exones.
El mecanismo en el que
intrones del grupo II se empalman (dos reacciones de transesterificación como
intrones del grupo I) es como sigue:
1. El 2'OH de un adenosina específico en el
intrón ataca el sitio 5 'de empalme, formando así el lazo
2. El 3'OH del exón 5 'desencadena la
transesterificación segundo en el extremo 3' del sitio de empalme con ello
unirse a los exones.
EMPALME tRNA
ARNt (tRNA-como también) de
empalme es otra forma rara de empalme que generalmente se presenta en el tRNA.
La reacción de empalme requiere una bioquímica diferente a las vías de
spliceosomal y auto-empalme. ribonucleasas escindir el ARN y ligasas unen los
exones.
SPLICING ALTERNATIVO
El splicing alternativo
(alternative splicing en inglés) o empalme alternativo permite obtener a partir
de un transcrito primario de mRNA o pre-ARNm distintas moléculas de mRNA
maduras. Este proceso ocurre principalmente en eucariotas, aunque también puede
observarse en virus.
Al transcribirse el ADN a
ARNm se obtiene un transcrito primario de ARN o pre-ARNm que incluye intrones y
exones. Para que este pre-ARNm de lugar a un ARNm debe sufrir un proceso de
maduración del ARNm, que consiste, básicamente, en eliminar todos los intrones.
Sin embargo los intrones y exones no siempre están determinados durante el
proceso de ayuste. La selección de los sitios de ayuste es llevado a cabo por
residuos de serina/arginina de ciertas proteínas conocidas como proteínas SR.
TIPOS DE SPLICING
ALTERNATIVOS:
(a) Selección de promotores
alternativos: este es el único método que da lugar a un dominio N-terminal
alternativo. En este caso, cada promotor puede dar lugar a un juego de exones
diferentes.
(b) Selección de sitios de
poliadenilación alternativos: este es el único método que da lugar a un dominio
C-terminalalternativo. En este caso, cada sitio de poliadenilación puede dar
lugar a un juego de exones diferentes.
(c) Retención de intrones:
en este caso en lugar de ayustar los intrones, estos son retenidos en el
transcrito. Este intrón puede expresarse, dar lugar a un codón de parada o
cambiar la pauta de lectura.
(d) Splicing de exones (exon
splicing): en este caso ciertos exones son sujetos a splicing fuera.
Bibliografia:
. http://es.wikipedia.org/wiki/Splicing_de_ARN





No hay comentarios:
Publicar un comentario